
Infectious Diseases & ARTIC Sequencing
Choose Type:
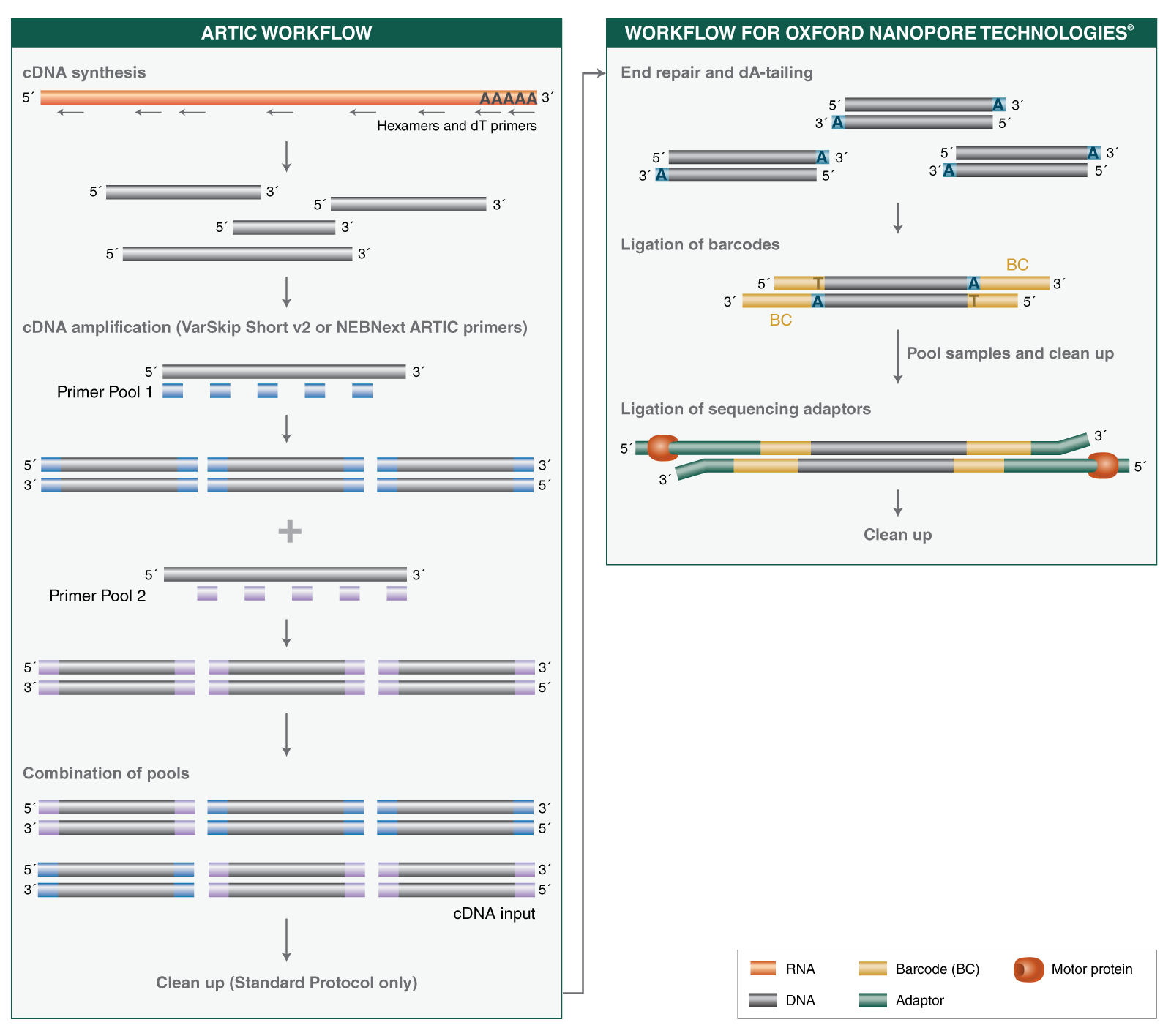
- Where can I find guidelines and protocols for using the NEBNext ARTIC SARS-CoV-2 RT-PCR Module, including cDNA synthesis and cDNA amplification?
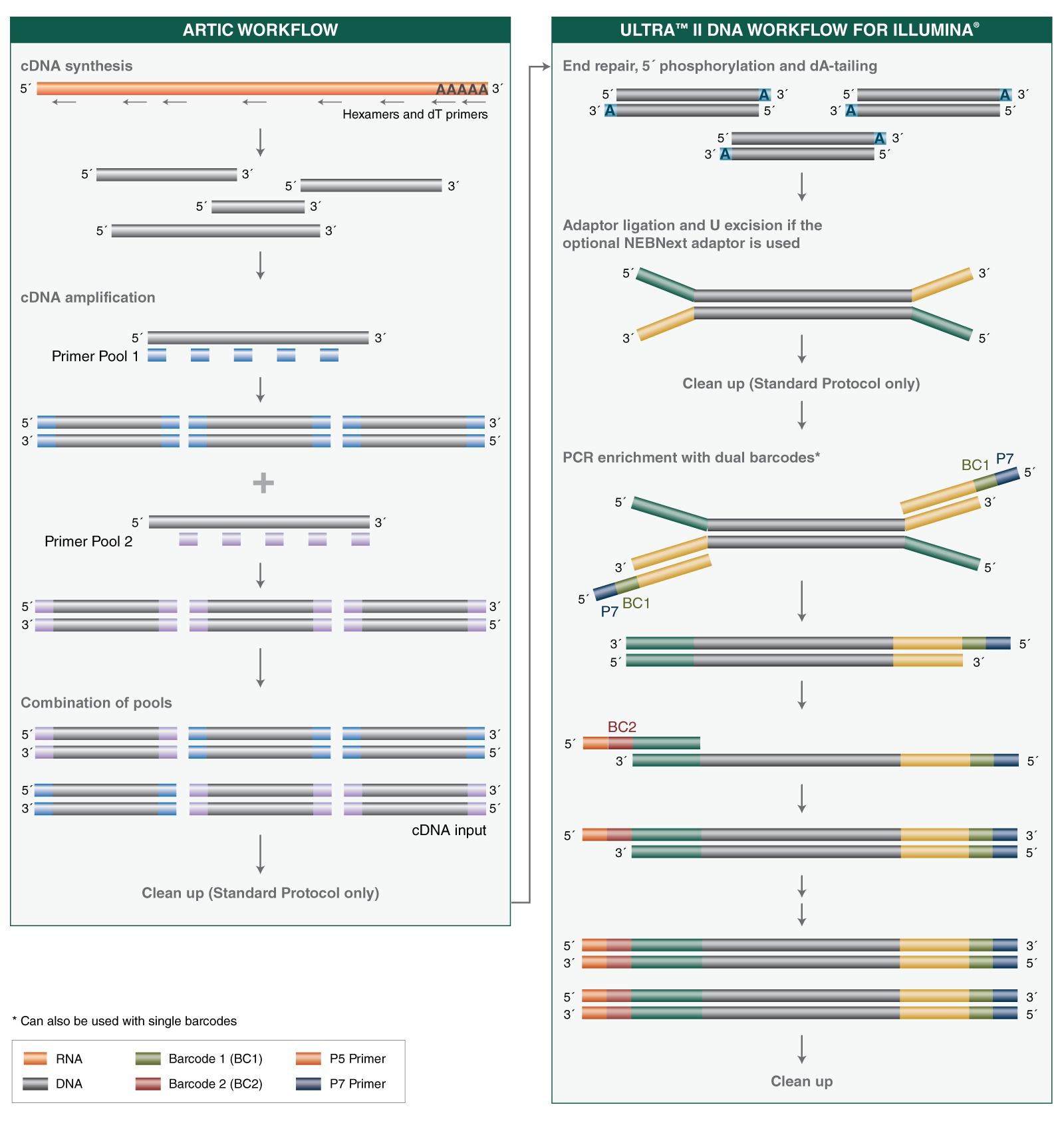
- Where can I find guidelines and protocols for using the NEBNext ARTIC SARS-CoV-2 Library Prep Kit, including cDNA synthesis, cDNA amplification and library prep?
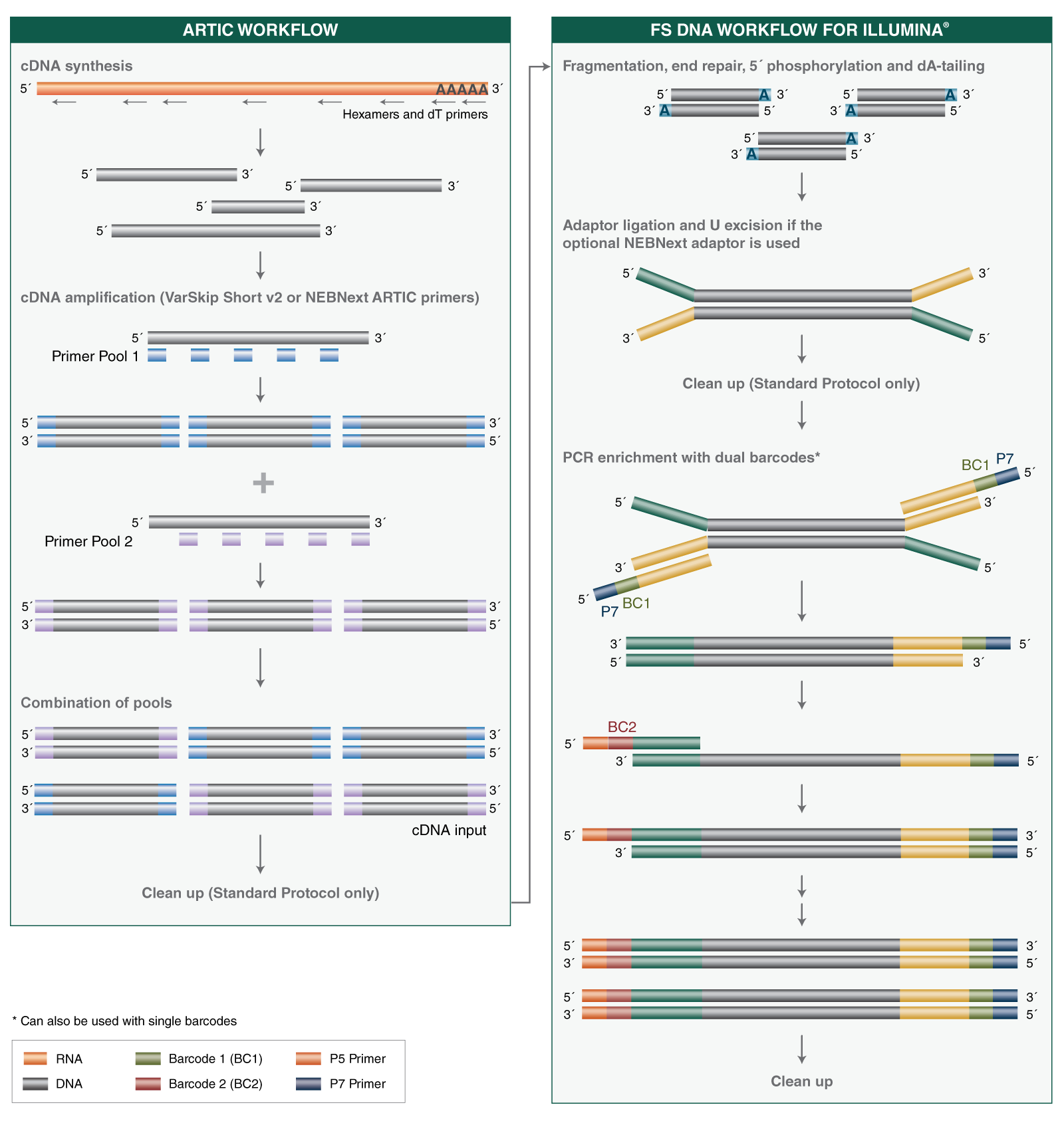
- Where can I find guidelines and protocols for using the NEBNext ARTIC SARS-CoV-2 FS Library Prep Kit, including cDNA synthesis, cDNA amplification and library prep, and the Express protocol option with reduced cleanup steps?
Products and content are covered by one or more patents, trademarks and/or copyrights owned or controlled by New England Biolabs, Inc (NEB). The use of trademark symbols does not necessarily indicate that the name is trademarked in the country where it is being read; it indicates where the content was originally developed. The use of this product may require the buyer to obtain additional third-party intellectual property rights for certain applications. For more information, please email busdev@neb.com.
This product is intended for research purposes only. This product is not intended to be used for therapeutic or diagnostic purposes in humans or animals.



